Search Nipponbare
| Mutant Information |
|---|
| Mutant Name | FN1423-S [Download] |
| Generation | M2 |
| Genus Species | Oryza Sativa |
| Cultivar | Kitaake |
| Alignment File (BAM File) | Download FN1423-S Alignment File |
| Seed Availability | Yes [Order Seeds] |
| Mapping (Hover to Zoom-In) | 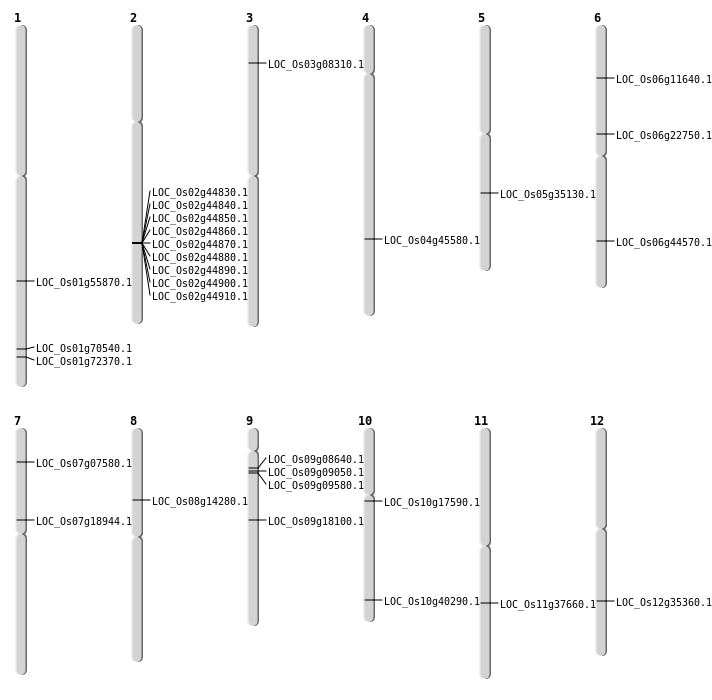
|
| Variant Information |
|---|
Single base substitutions: 118
| Variant Type | Chromosome | Position | Change | Genotype | Effect | Gene Affected |
|---|---|---|---|---|---|---|
| SBS | Chr1 | 21847884 | G-A | HOMO | SYNONYMOUS_CODING | |
| SBS | Chr1 | 23742726 | C-A | HOMO | INTRON | |
| SBS | Chr1 | 24921674 | G-T | HOMO | INTRON | |
| SBS | Chr1 | 25001002 | A-T | HOMO | INTRON | |
| SBS | Chr1 | 29717765 | G-C | HET | INTERGENIC | |
| SBS | Chr1 | 30318159 | C-T | HET | INTERGENIC | |
| SBS | Chr1 | 30450708 | T-C | HET | INTERGENIC | |
| SBS | Chr1 | 32095660 | T-A | HET | INTRON | |
| SBS | Chr1 | 36218892 | T-C | HET | INTERGENIC | |
| SBS | Chr1 | 37876072 | G-T | HET | INTERGENIC | |
| SBS | Chr1 | 37876075 | C-A | HET | INTERGENIC | |
| SBS | Chr1 | 5985911 | T-G | HOMO | INTERGENIC | |
| SBS | Chr10 | 14044873 | A-T | HOMO | INTERGENIC | |
| SBS | Chr10 | 21595770 | G-T | HET | NON_SYNONYMOUS_CODING | LOC_Os10g40290 |
| SBS | Chr10 | 8883050 | A-G | HOMO | NON_SYNONYMOUS_CODING | LOC_Os10g17590 |
| SBS | Chr10 | 917972 | G-A | HOMO | INTERGENIC | |
| SBS | Chr10 | 917973 | C-A | HOMO | INTERGENIC | |
| SBS | Chr11 | 15618770 | C-G | HOMO | INTERGENIC | |
| SBS | Chr11 | 18233913 | G-T | HET | INTERGENIC | |
| SBS | Chr11 | 19474888 | G-T | HET | INTERGENIC | |
| SBS | Chr11 | 22230163 | C-G | HET | NON_SYNONYMOUS_CODING | LOC_Os11g37660 |
| SBS | Chr11 | 23882804 | G-C | HET | SYNONYMOUS_CODING | |
| SBS | Chr11 | 26824125 | G-C | HET | INTRON | |
| SBS | Chr11 | 26996234 | G-C | HET | INTERGENIC | |
| SBS | Chr11 | 27627847 | C-A | HET | INTERGENIC | |
| SBS | Chr11 | 5765098 | C-G | HET | INTERGENIC | |
| SBS | Chr11 | 7969526 | G-A | HOMO | INTERGENIC | |
| SBS | Chr12 | 14914206 | C-A | HOMO | INTRON | |
| SBS | Chr12 | 15075812 | C-A | HOMO | INTERGENIC | |
| SBS | Chr12 | 15500269 | G-A | HOMO | INTERGENIC | |
| SBS | Chr12 | 17697037 | G-T | HET | INTRON | |
| SBS | Chr12 | 17697040 | G-T | HET | INTRON | |
| SBS | Chr12 | 23626467 | C-T | HOMO | INTERGENIC | |
| SBS | Chr12 | 24310352 | G-T | HOMO | INTERGENIC | |
| SBS | Chr12 | 2486570 | C-T | HET | INTERGENIC | |
| SBS | Chr12 | 26063493 | C-A | HET | INTERGENIC | |
| SBS | Chr12 | 7239456 | C-A | HET | INTERGENIC | |
| SBS | Chr2 | 10299166 | G-A | HET | INTRON | |
| SBS | Chr2 | 11066022 | G-A | HET | INTERGENIC | |
| SBS | Chr2 | 11658179 | T-A | HET | INTERGENIC | |
| SBS | Chr2 | 14545644 | C-T | HOMO | INTERGENIC | |
| SBS | Chr2 | 14986451 | C-T | HOMO | INTERGENIC | |
| SBS | Chr2 | 25748882 | T-A | HET | INTERGENIC | |
| SBS | Chr2 | 25748883 | C-A | HET | INTERGENIC | |
| SBS | Chr2 | 34717389 | G-A | HOMO | INTERGENIC | |
| SBS | Chr2 | 4072992 | C-T | HOMO | INTERGENIC | |
| SBS | Chr3 | 10531968 | A-T | HOMO | INTERGENIC | |
| SBS | Chr3 | 11092757 | A-G | HOMO | INTERGENIC | |
| SBS | Chr3 | 13962190 | G-A | HOMO | INTRON | |
| SBS | Chr3 | 14500839 | C-A | HOMO | INTERGENIC | |
| SBS | Chr3 | 1754340 | C-A | HOMO | INTRON | |
| SBS | Chr3 | 17557923 | G-A | HOMO | INTERGENIC | |
| SBS | Chr3 | 18077755 | T-A | HOMO | INTERGENIC | |
| SBS | Chr3 | 22032938 | C-A | HOMO | INTERGENIC | |
| SBS | Chr3 | 29118687 | G-A | HET | INTRON | |
| SBS | Chr3 | 31490524 | C-T | HET | SYNONYMOUS_CODING | |
| SBS | Chr3 | 33123128 | C-T | HET | INTERGENIC | |
| SBS | Chr3 | 34534476 | C-T | HOMO | INTERGENIC | |
| SBS | Chr3 | 35945262 | T-A | HET | INTERGENIC | |
| SBS | Chr3 | 4232690 | G-T | HOMO | STOP_GAINED | LOC_Os03g08310 |
| SBS | Chr3 | 6811747 | C-G | HOMO | INTERGENIC | |
| SBS | Chr3 | 9055667 | G-T | HOMO | INTERGENIC | |
| SBS | Chr4 | 11835904 | C-T | HOMO | INTERGENIC | |
| SBS | Chr4 | 2251212 | G-T | HET | INTRON | |
| SBS | Chr4 | 6361591 | G-A | HOMO | SYNONYMOUS_CODING | |
| SBS | Chr5 | 14554857 | C-G | HET | INTERGENIC | |
| SBS | Chr5 | 1799167 | C-A | HOMO | INTERGENIC | |
| SBS | Chr5 | 19591401 | G-A | HET | INTERGENIC | |
| SBS | Chr5 | 20867876 | G-A | HET | NON_SYNONYMOUS_CODING | LOC_Os05g35130 |
| SBS | Chr5 | 20980742 | C-G | HET | INTERGENIC | |
| SBS | Chr5 | 21828771 | G-T | HET | INTERGENIC | |
| SBS | Chr5 | 22688925 | C-T | HET | INTERGENIC | |
| SBS | Chr5 | 26614646 | G-A | HET | INTERGENIC | |
| SBS | Chr5 | 28477820 | C-A | HET | INTERGENIC | |
| SBS | Chr5 | 6405161 | C-T | HET | INTERGENIC | |
| SBS | Chr5 | 815971 | T-C | HOMO | INTERGENIC | |
| SBS | Chr5 | 815979 | T-C | HOMO | INTERGENIC | |
| SBS | Chr6 | 12239577 | T-A | HOMO | INTRON | |
| SBS | Chr6 | 12239578 | C-A | HOMO | INTRON | |
| SBS | Chr6 | 13718718 | T-C | HOMO | INTRON | |
| SBS | Chr6 | 21893671 | A-G | HOMO | INTRON | |
| SBS | Chr6 | 23865116 | G-C | HET | INTERGENIC | |
| SBS | Chr6 | 26916819 | G-T | HET | NON_SYNONYMOUS_CODING | LOC_Os06g44570 |
| SBS | Chr6 | 29091126 | G-A | HET | INTERGENIC | |
| SBS | Chr6 | 3572932 | C-T | HET | INTERGENIC | |
| SBS | Chr6 | 6181979 | C-G | HET | INTERGENIC | |
| SBS | Chr6 | 6523523 | T-C | HET | INTERGENIC | |
| SBS | Chr7 | 10281568 | G-A | HOMO | INTERGENIC | |
| SBS | Chr7 | 11040415 | C-G | HET | INTERGENIC | |
| SBS | Chr7 | 11048634 | C-T | HOMO | INTERGENIC | |
| SBS | Chr7 | 11048637 | G-A | HOMO | INTERGENIC | |
| SBS | Chr7 | 11048638 | G-T | HOMO | INTERGENIC | |
| SBS | Chr7 | 16636548 | T-A | HOMO | INTERGENIC | |
| SBS | Chr7 | 17502061 | C-T | HOMO | INTRON | |
| SBS | Chr7 | 17547318 | C-A | HOMO | INTERGENIC | |
| SBS | Chr7 | 19815076 | G-A | HOMO | INTRON | |
| SBS | Chr7 | 25760216 | G-A | HOMO | INTERGENIC | |
| SBS | Chr7 | 29683820 | A-T | HOMO | INTERGENIC | |
| SBS | Chr7 | 3805511 | C-T | HOMO | INTRON | |
| SBS | Chr7 | 7054926 | C-A | HOMO | INTERGENIC | |
| SBS | Chr8 | 23869266 | T-C | HET | INTERGENIC | |
| SBS | Chr8 | 24544420 | G-A | HET | INTRON | |
| SBS | Chr8 | 26520131 | C-G | HET | INTRON | |
| SBS | Chr8 | 8248578 | A-G | HOMO | INTERGENIC | |
| SBS | Chr8 | 843792 | A-G | HOMO | INTERGENIC | |
| SBS | Chr8 | 8557158 | C-T | HET | NON_SYNONYMOUS_CODING | LOC_Os08g14280 |
| SBS | Chr9 | 11095881 | G-T | HET | SYNONYMOUS_CODING | |
| SBS | Chr9 | 11095882 | C-T | HET | NON_SYNONYMOUS_CODING | LOC_Os09g18100 |
| SBS | Chr9 | 1166028 | C-A | HET | INTRON | |
| SBS | Chr9 | 12983218 | C-T | HET | INTERGENIC | |
| SBS | Chr9 | 15381501 | C-T | HET | INTERGENIC | |
| SBS | Chr9 | 17627418 | C-G | HET | INTRON | |
| SBS | Chr9 | 18738037 | G-T | HET | INTRON | |
| SBS | Chr9 | 1963069 | C-T | HET | INTRON | |
| SBS | Chr9 | 4517496 | G-A | HET | NON_SYNONYMOUS_CODING | LOC_Os09g08640 |
| SBS | Chr9 | 4825158 | G-C | HET | NON_SYNONYMOUS_CODING | LOC_Os09g09050 |
| SBS | Chr9 | 5167579 | C-A | HET | STOP_GAINED | LOC_Os09g09580 |
| SBS | Chr9 | 9709317 | A-G | HET | INTERGENIC |
Deletions: 15
| Variant Type | Chromosome | Start | End | Size (bp) | Number of Genes |
|---|---|---|---|---|---|
| Deletion | Chr7 | 3798393 | 3798404 | 11 | LOC_Os07g07580 |
| Deletion | Chr8 | 6031120 | 6031126 | 6 | |
| Deletion | Chr6 | 6181788 | 6181789 | 1 | |
| Deletion | Chr5 | 7908379 | 7908380 | 1 | |
| Deletion | Chr7 | 9423356 | 9423359 | 3 | |
| Deletion | Chr7 | 11048081 | 11048083 | 2 | |
| Deletion | Chr6 | 13233037 | 13233046 | 9 | LOC_Os06g22750 |
| Deletion | Chr1 | 16141241 | 16141246 | 5 | |
| Deletion | Chr8 | 22208251 | 22208252 | 1 | |
| Deletion | Chr1 | 22265823 | 22265824 | 1 | |
| Deletion | Chr12 | 22624612 | 22624627 | 15 | |
| Deletion | Chr4 | 26962594 | 26962604 | 10 | LOC_Os04g45580 |
| Deletion | Chr2 | 27141001 | 27209000 | 68000 | 9 |
| Deletion | Chr4 | 28438484 | 28438485 | 1 | |
| Deletion | Chr1 | 40857059 | 40857069 | 10 | LOC_Os01g70540 |
Insertions: 1
| Variant Type | Chromosome | Start | End | Size (bp) | Number of Genes |
|---|---|---|---|---|---|
| Insertion | Chr9 | 5029682 | 5029683 | 2 |
Inversions: 2
| Variant Type | Chromosome | Position 1 | Position 2 | Number of Genes |
|---|---|---|---|---|
| Inversion | Chr8 | 6089459 | 6528946 | |
| Inversion | Chr1 | 32172605 | 41977140 | 2 |
Translocations: 6
| Variant Type | Chromosome | Position 1 | Chromosome | Position 2 | Number of Genes |
|---|---|---|---|---|---|
| Translocation | Chr12 | 2322097 | Chr11 | 5019906 | |
| Translocation | Chr7 | 11222735 | Chr4 | 17934859 | 2 |
| Translocation | Chr11 | 13220820 | Chr5 | 29713157 | |
| Translocation | Chr12 | 21494753 | Chr11 | 17511063 | LOC_Os12g35360 |
| Translocation | Chr11 | 23704834 | Chr6 | 6173489 | LOC_Os06g11640 |
| Translocation | Chr11 | 23704841 | Chr6 | 6173498 | LOC_Os06g11640 |
